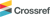홉 추출물 성분프로파일 및 지표물질 분리
Abstract
Humulus lupulus (hops) is a hermaphroditic plant with male and female flowers occurring in separate individuals. The ethanolic extract of hops, the female flower cones, was analyzed by LC/MS-based molecular networking (MN) approach to selectively isolate xanthohumol (1), xanthohumol D (2), xanthohumol B (3), and isoquercitrin (4) with therapeutic properties such as antimicrobial, anti-inflammatory, and antioxidant effects. Hops were extracted with 50% ethanol and 1 was sequentially isolated using various chromatographic methods. From the EtOAc fraction containing 1 identified by MN, 1 was isolated using column chromatography and characterized by LCMS and identified by NMR analyses. We also profiled other specialized metabolites of H. lupulus by visualizing MN.
Keywords:
Humulus lupulus, olecular networking (MN), Xanthohumol홉(Humulus lupulus)은 삼과(Cannabaceae)에 속하는 유럽 및 북미 원산의 덩굴성 초본 다년생식물으로 암꽃을 주로 의약적, 산업적 용도로 이용되고 있다. 특히, 양조산업에서 맥주의 쓴맛과 향을 더하는데 사용된다.1,2) 홉의 성분으로는 prenylflavonoid 계열인 xanthohumol을 비롯하여 isoxanthohumol, 8-prenylnarigenin, 6-prenylnaringenin, humulone (α-acid), lupulone (β-acid), isoquercitrin 등이 있다.2,3) Xanthohumol은 홉의 주성분으로 관련 prenylchalcone는 항암효과가 있다고 알려져 있으며, 이외의 flavonoids는 항산화, 대사장애 조절, 진정작용, 항염증과 같은 활성효과가 있고 알려져 있다.2,3)
MS 기기의 발전으로 데이터 처리속도, 민감도, 분해능이 증가하면서 생성되는 데이터의 양과 복잡성이 늘어남에 따라서 복잡한 데이터를 처리하기 위해 LC-MS같은 프로파일링 과정이 대사체학 연구에서 적용된다.4,5) 분자 네트워킹(molecular networking)은 비표적 질량분석(MS) 데이터를 시각화하여 주석(annotate)하는 생물정보학 핵심 기법이다.4) MZmine은 2005년부터 개발된 프로그램으로 LC-MS, GC-MS, IMS-MS 및 MS 데이터를 통합하여 분석할 수 있으며 대사체학 연구에 활용된다.4,5) MZmine은 측정된 질량분석 데이터의 시간(RT), 질량(m/z), 이온 이동도(ion mobility)를 정렬하고 “Recursive threshold” 알고리즘을 통해 noise로 분류되는 peak를 제거하여 신뢰성을 높인다.3,4) MZmine을 통해서 처리된 데이터는 SIRIUS, MetaboAnalyst, GNPS 등 다른 분석 플랫폼과 연계되어 사용된다.4) Global Natural Products Social Molecular Networking (GNPS)는 데이터를 시각화 하기 위해 사용되는 플랫폼으로 MS2 (tandem 질량 분석)데이터를 활용하여 스펙트럼 유사도에 따라 관련 분자를 연결한다.5) GNPS 플랫폼은 질량 분석 기반 대사체학 연구에 활용되며 GNPS내의 다양한 workflow를 통해 분자네트워킹에 사용된다.5)
홉은 여러 효능가치가 보고된 식물로 미래에 생물학적 응용이 가능할 것으로 보여지는 식물로 연구가치가 충분하다고 판단된다. 본 연구에서는 고성능액체크로마토그래피(HPLC)와 질량분석법(MS)을 통해 얻은 질량분석 데이터로 분자네트워크 접근법을 이용하여 홉에 다양하게 분포 되어있는 미량성분들을 분석하였다. 분자네트워크 접근법을 활용하여 3종의 chalcone과 1종의 flavonol을 분리하고 구조를 동정하였다.
재료 및 방법
실험 재료 -실험에 사용한 홉 추출물(49 g)은 (주)뉴트리케어를 통해 50% 주정 EtOH로 추출된 분말 형태로 제공받았다.
기기 및 시약 –분석용 고성능 액체크로마토그래피(Ultra-High performance liquid chromatography, UHPLC)는 Thermo사의 UHPLC pump, UHPLC detector가 장착된 Vanquish Flex-A10 UHPLC를 사용하였다. 고분해능 질량분석장비는 Thermo사의 Orbitrap Exploris 120을 사용하였고, UHPLC-MS column (Hypersil GOLDTM aQ, 5 µm, 100*2.1 mm)을 사용하였다. TLC 분석은 Merck사의 TLC-silica gel: 60 F254 and RP-18 F254 plates를 사용하였다. 중압액체크로마토그래피(MPLC)는 CombiFlash®PF+ (Teledyne Isco, USA)와 RediSep®사의 NP silica gel, NP column을 사용하였다. 분취용 HPLC는 PDA detector를 구비된 Waters사(Milford, MA, USA)의 996을 사용하였으며,
분취용 컬럼은 YMC사의 actus Triart C18 (250*20 mm I.D.S-5 µm, 12 nm; Seongnam-si, Gyeonggi-do, Republic of Korea)을 사용하였다. 1H-NMR (500 MHz)과 13C-NMR spectra (125 MHz)는 강원대학교 항암혁신신약개발 핵심연구지원센터에 설치된 JEOL JNM-ECZR 500Mhz 기기를 사용하였다. Sephadex는 LH-20 (18-111 um, GE Healthcare, Sweden)를 사용하였다. HPLC용매는 MeOH, MeOH, can (Honeywell, Charlotte, North Carolina, USA)을 사용하였다. 기타 유기용매는 1-utanol (삼전순약공업(주), Seoul, Korea), DCM (삼전순약공업(주), Seoul, Korea), n-exane (삼전순약공업(주), Seoul, Korea)을 사용하였다. NMR 측정 용매는 Cambridge Isotope Laboratories (Tewksbury, MA, USA)의 methanol-d4 (CD3OD), dimethyl sulfoxide-d6 (DMSO-d6)을 사용하였다.
추출 및 분리 –뉴트리케어로부터 50% 주정EtOH로 추출된 홉 49 g을 받아 진행하였다. 농축되어 있는 추출물을 증류수로 현탁하여 n-hexane, EtOAc, n-BuOH로 순차적으로 분획한 뒤 감압 농축하여 각각 1.86, 8.68, 6.58 g을 획득하였다. EtOAc 분획물 (8.68 g)을 순상 컬럼인 RediSep® NP silica gel에 n-Hex-EtOAc=7:3, 6:4, 5:5, 4:6, 3:7과 DCM-MeOH=9:1, 8:2, 7:3을 용매로 순차적으로 중압액체크로마토그래피를 진행하였고, TLC분석을 실시, 18개의 하위 분획물(HLE-1 ~ 18)로 나누었으며, 화합물 4 (255.1 mg)를 얻었다. HLE-5 분획물을 Sephadex LH-20을 고정상으로 하고 MeOH를 이동상으로 한 open column chromatography를 실시하여 용출시켜 12개의 소분획과 화합물 1 (50.4 mg)을 얻었다(HLE5-1~HLE5-12). HLE5-9를 분취용 HPLC를 이용해 [MeOH:H2O (8:2), 5mL/min]조건으로 이동상을 용출시켜 화합물 2 (1.6 mg) 및 3 (1.9 mg)을 얻었다.
분자 네트워크법 홉 추출물의 하위 분획물(HLE5-9, HLE16)의 raw 데이터를 mzmine 소프트웨어(version. 3.9.0 august 22 2023)을 사용하였으며, noise를 제거하기 위해 ms detection ms noise level MS1과 MS2은 각각 MS 1: 1.0 E5 MS 2: 6.0 E3으로 설정하였으며, parameter setting은 ADAP chromatogram builder를 사용하였으며, min height intensity: 1.0 E6, m/z tolerance: 0.0030 m/z or 5.0 ppm으로 설정하여 mgf file로 변환 후 GNPS (https://gnps2.org/homepage)의 분자네트워크 웹사이트를 통해 처리하였다. workspace는 feature-based molecular networking (FBMN)을 사용하였다. precursor ion tolerance 및 fragment ion mass tolerance는 0.02 Da 로 설정하였으며, minimum cosine score 0.7, min matched peaks 6개이다. MS스펙트럼 데이터는 GNPS 스펙트럼 라이브러리에서 검색되었으며, MN결과를 시각화하기 위해 cytoscape (version. 3.10.2)를 사용하여 진행하였다.
Xanthohumol (1) – yellow powder; 1H NMR (500 MHz, DMSO-d6) δ 14.62 (s, 1H), 7.74 (d, J=15.5 Hz, 1H, H-α), 7.65 (d, J=15.6 Hz, 1H, H-β), 7.55 (d, J=8.6 Hz, 2H, H-2,6), 6.82 (d, J=8.6 Hz, 2H, H-3,5), 6.05 (s, 1H, H-5'), 5.12 (m, 1H, H-1''), 3.84 (s, 3H, 6'-OCH3), 3.12 (d, J=7.1 Hz, 2H, H-2''), 1.68 (s, 3H, H-5''), 1.59 (s, 3H, H-4''); 13C NMR (125 MHz, DMSO-d6) δ 191.6 (ketone), 164.63 (C-2'), 160.55 (C-6'), 159.93 (C-4), 142.5 (C-β), 130.5 (C-2,6), 129.9 (C-3''), 126.1 (C-1), 123.8 (C-α), 123.1 (C-2''), 116.0 (C-3,5), 107.4 (C-3'), 104.5 (C-1'), 91.05 (C-5'), 55.8 (C-6'-OCH3), 25.5 (C-4''), 21.1 (C-1''), 17.7 (C-5''); ESI-MS m/z 355.1538 [M+H]+.
Xanthohumol D (2) – Pale yellow powder; 1H NMR (500 MHz, methanol-d4): δ 7.82 (d, J=15.5 Hz, 1H, H-α), 7.70 (d, J=15.5 Hz, 1H, H-β), 7.53 (d, J=8.5 Hz, 2H, H-2,6), 6.85 (d, J=8.6 Hz, 2H, H-3,5), 6.06 (s, 1H, H-5'), 4.83 (m, 1H, H-1''a), 4.73 (m, 1H, H-1''b), 4.36 (m, 1H, H--2''), 3.93 (s, 3H, 6'-OCH3), 2.98 (dd, J=13.8, 5.5 Hz, 1H, H-4''a), 2.81 (dd, J=13.8, 7.4 Hz, 1H, H-4''b), 1.83 (s, 3H, H-5''); 13C NMR (125 MHz, methanol-d4): δ 194.1 (ketone), 166.6 (C-6'), 165.2 (C-4'), 162.9 (C-2'), 161.1 (C-4), 148.9 (C-3''), 143.5 (C-β), 131.3 (C-2,6), 128.5 (C-α), 125.8(C-1), 116.9 (C-3,5), 110.8 (C-4''), 106.8 (C-5'), 106.2 (C-1'), 92.2 (C-3'), 76.8 (C-2''), 56.2 (C-6'-OCH3), 29.8 (C-1''), 17.9 (C-5''); ESI-MS m/z 371.1487 [M+H]+.
Xanthohumol B (3) – Pale yellow powder; 1H NMR (500 MHz, methanol-d4) δ 7.84 (d, J=15.5 Hz, 1H, H-α), 7.73 (d, J=15.6 Hz, 1H, H-β), 7.53 (d, J=8.5 Hz, 2H, H-2,6), 6.85 (d, J=8.6 Hz, 2H, H-3,5), 6.02 (s, 1H, H-5'), 3.93 (s, 3H, 6'-OCH3), 3.79 (dd, J= 6.9, 5.3 Hz, 1H, H-2''), 2.87 (dd, J=16.7, 5.3 Hz, 1H, H-1''a), 2.55 (dd, J=16.7, 6.9 Hz, 1H, H-1''b), 1.37 (s, 3H, H-4''), 1.32 (s, 3H, H-5''); 13C NMR (125 MHz, methanol-d4) δ 194.2 (ketone), 166.3 (C-2'), 162.5 (C-6'), 161.3 (C-4'), 143.9 (C-β), 131.4 (C-2,6), 128.4 (C-1), 125.5 (C-α), 116.9 (C-3,5), 106.7 (C-3'), 101.9 (C-1'), 92.9 (C-5'), 79.71 (C-3''), 69.9 (C-2''), 56.3 (C-6'-OCH3), 26.2 (C-1''), 25.7 (C-4''), 21.4 (C-5''); ESI-MS m/z 371.1490 [M+H]+.
Isoquercitrin (4) – Dark yellow powder; 1H-NMR (500 MHz, methanol-d4) δ 7.73 (d, J=2.2 Hz, 1H, H-2'), 7.62 (dd, J=8.5, 2.2 Hz, 1H, H-6'), 6.90 (d, J=8.5 Hz, 1H, H-5'), 6.43 (d, J=2.1 Hz, 1H, H-8), 6.24 (d, J=2.1 Hz, 1H, H-6), 5.28 (d, J=7.6 Hz, 1H, H-1''), 3.74 (dd, J=11.9, 2.4 Hz, 1H, H-6''a), 3.60 (dd, J=11.9, 5.4 Hz, 1H, H-6''b), 3.51 (dd, J=9.1, 7.6 Hz, 1H, H-3''), 3.45 (t, J=8.9 Hz, 1H, H-2''), 3.37 (m, 1H, H-4''), 3.24 (m, J=9.6, 5.4, 2.4 Hz, 1H, H-5''); 13C NMR (125 MHz, methanol-d4) δ 179.5 (C-4), 166.1 (C-7), 163.1 (C-5), 159.0 (C-2), 158.5 (C-9), 149.9 (C-4'), 145.9 (C-5'), 135.6 (C-3), 123.2 (C-5'), 123.1 (C-1'), 105.7 (C-10), 104.3 (C-1''), 99.9 (C-6), 94.7 (C-8), 78.4 (C-5''), 78.1 (C-3''), 75.7 (C-2''), 71.2 (C-4''), 62.6 (C-6''); ESI-MS m/z 487.0848 [M+Na]+.
결과 및 고찰
화합물 1의 HRESI-MS 스펙트럼에서 m/z 355.1538 [M+H]+를 통해 분자식은 C21H22O5임을 유추할 수 있었다. 화합물 1은 분자 네트워크법을 이용하여 MS/MS pattern을 GNPS library와 비교하고 참고문헌을 비교해 chalcone 구조이며, xanthohumol임을 유추하였다(Fig. 1 and 2).7) 1의 1H NMR 스펙트럼의 δH 7.56 (2H, d, J=8.6 Hz, H-2,3), 6.83 (2H, d, J=8.6 Hz, H-5,6)에서 나타나는 doublet이 벤젠고리의 4-치환체를 유추하였으며, δH 7.75 (1H, d, J=15.5 Hz, H-α), 7.65 (1H, d, J=15.5 Hz, H-β) 신호로 trans 형태의 이중결합을 갖고 있음을 확인하였고, δH 3.85 (3H, s, 6'-OCH3)으로 벤젠고리에 메틸기가 있음을 확인하였고, 13C NMR의 결과로 케톤기(δC 191.6)를 갖고 있음을 확인하여, 이상의 결과로 화합물 1은 chalcone구조임을 유추하였다. 1H-NMR에서 singlet 형태의 δH 1.68 (s, 3H, H-5''), 1.59 (s, 3H, H-4'')으로 2개의 메틸기와 δH 3.12 (d, J=7.1 Hz, 2H, H-2'')과 5.12 (m, 1H, H-1'')의 coupling constant는 각각 doublet, multiplet을 확인하여 prenyl기가 있음을 유추하였고, 이러한 결과로 화합물 1의 구조를 분석자료를 참고문헌과 비교하여 xanthohumol로 동정하였다(Fig. 2).8)
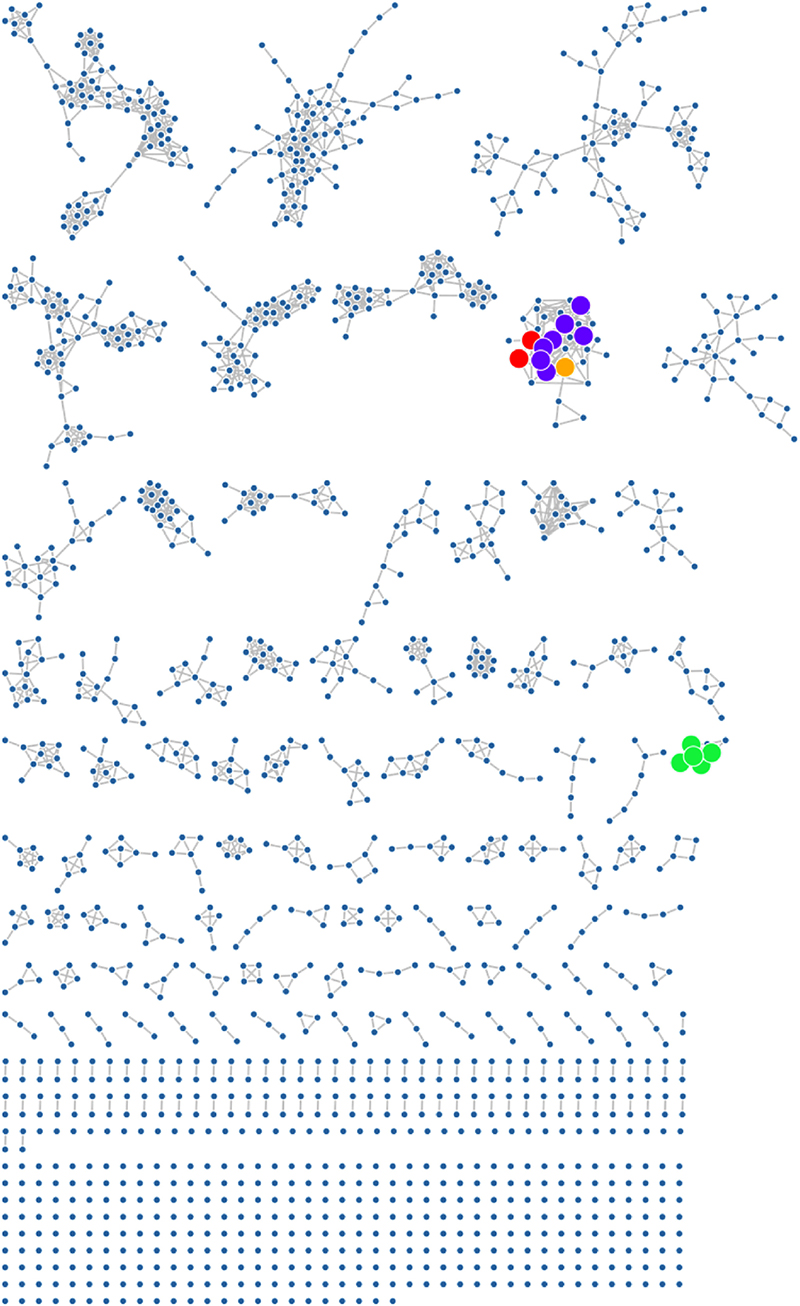
Molecular network of the total extract from H. lupulus. Node colors indicate the following: purple (1), red (2), orange (3), and green (4), corresponding to compounds 1–4, respectively.
화합물 2는 HRESI-MS 스펙트럼에서 m/z 371.1487 [M+H]+를 통해 C21H22O6로 보아 화합물 1에 알코올기가 추가된 것을 유추할 수 있었다. 화합물 2는 분자네트워크법을 통해 1과 같은 cluster에 있으며, MS/MS pattern을 참고문헌과 비교하여 1의 유도체임을 유추하였다(Fig. 1).9) 화합물 2의 1H-NMR 스펙트럼 δH 4.83 (H, m, J=1.05 Hz, H-1''a), 4.73 (H, m, J=1.05 Hz, H-1''b)의 신호로 2'' 위치에 1과 달리 치환체를 갖고 있음을 알 수 있었고, 13C-NMR을 통해 치환체로 알코올기가 결합되어 있음을 알 수 있었다. δH 1.83 (s, 3H, H-5'')하나의 메틸기와 δH 2.98 (H, dd, 13.8, J=5.5 Hz, H-4''), 2.81 (H, dd, J=13.8, 7.4 Hz, H-4'')의 신호로 geminal 형태의 이중결합을 이루고 있음을 유추하였고, HMBC를 통해 H-5''이 C-2'',3'',4''에 correlation peak (δH 1.83/δC 76.8, 148.9, 110.8) 함을 통해 C-3''위치에 메틸기가 있음으로 2''위치에 알코올기가 있는 isoprenyl기임을 유추하였다. 이러한 결과로 화합물 2의 구조를 분석자료를 참고문헌과 비교하여 xanthohumol D로 동정하였다(Fig. 2)8)
화합물 3는 HRESI-MS 스펙트럼에서 m/z 371.1487 [M+H]+를 통해 C21H22O6으로 2의 유도체로 예상하였다. 화합물 3은 2와 달리 다른 클러스터에 있으나, 분자 네트워크법으로 MS/MS pattern을 참고문헌과 비교하여 2의 유도체임을 유추하였다(Fig. 1).10) δH 2.87 (H, dd, J=16.75, 5.33 Hz, H-1''a), 2.55 (H, dd, J=16.74, 6.94 Hz, H-1''b)으로 2''이 치환체를 갖고 있음을 유추하였다. 13C-NMR δC 69.9 (C-2'')신호로 2''위치에 알코올기가 연결되어 있고, δC 79.7 (C-3'')신호로 exomethylene 구조를 가진 화합물 2와 달리 chromane 골격을 유추하였다. 또한 2개의 메틸기 신호 [δH 1.37 (s, 3H, H-4''), 1.32 (s, 3H, H-5'')]를 확인하였고, HMBC 스펙트럼을 통해 H-4'', H-5''과 C-3''의 correlation (δH 1.37, 1.32/δC 75.1, 79.7)을 확인하여 두 개의 메틸기가 모두 3'' 위치에 결합된 것을 확인하였다. 이러한 결과로 화합물 3을 참고문헌과 비교한 결과 xanthohumol B로 동정하였다(Fig. 2).8)
화합물 4는 분리과정 중에 추가로 분리된 화합물로서, NMR 정보를 통해 chalchone 유도체가 아님을 확인하였다.11) 화합물 4의 HRESI-MS 스펙트럼에서 m/z 465.1026 [M+H]+를 통해 분자식은 C21H21O12 임을 유추하였다. 분자네트워크법을 통해 flavonoids 유도체 중 isoquercitrin을 추측하였고, 참고문헌과 비교하여 그 값과 1H와 13C-NMR 스펙트럼을 통해 isoquercitrin으로 동정하였다(Fig. 2).12) 또한 분리한 화합물을 표준품으로 이용하여, 검출시간과 MS/MS 데이터를 비교하여 분자네트워크 내에서 해당 물질의 노드를 확인할 수 있었으며, chalcone 유도체와는 다른 cluster에 존재하고 있음을 확인하였다(Fig. 1)
결 론
홉의 주정 EtOH 추출물로 분리한 chalcone 3종과 flavonoid 1종을 분리하였다. 분자네트워크법을 통해 형성된 cluster를 이용하여 xanthohumol (1) xanthohumol D (2), xanthohumol B (3) 및 isoquercitrin (4)을 분리하고, 분광학 분석을 통해 동정하였다.
Acknowledgments
본 연구는 농촌진흥청의 농업과학기술 공동연구사업(과제번호: RS-2022-RD010295)에 의해 수행한 결과이며, 또한 핵자기공명분광기 사용은 강원대학교 항암혁신신약개발 핵심연구지원센터(CFICDD)의 기술적 지원으로 수행되었으며, 지원에 감사드립니다.
References
-
Stevens, J. F. and Page, J. E. (2004) Xanthohumol and related prenylflavonoids from hops and beer: to your good health. Phytochemistry 65: 1317-1330.
[https://doi.org/10.1016/j.phytochem.2004.04.025]
-
Stevens, J. F., Taylor, A. W. and Deinzer, M. L. (1999) Quantitative analysis of xanthohumol and related prenylflavonoids in hops and beer by liquid chromatography–tandem mass spectrometry. J. Chromatogr. A. 832: 97-107.
[https://doi.org/10.1016/S0021-9673(98)01001-2]
-
Nookandeh, A., Frank, N., Steiner, F., Ellinger, R., Schneider, B., Gerhäuser, C. and Becker, H. (2004) Xanthohumol metabolites in faeces of rats. Phytochemistry 65: 561-5706.
[https://doi.org/10.1016/j.phytochem.2003.11.016]
-
Pluskal, T., Castillo, S., Villar-Briones, A. and Orešič, (2010) M. MZmine 2: Modular framework for processing, visualizing, and analyzing mass spectrometry-based molecular profile data. BMC Bioinform 11: 395.
[https://doi.org/10.1186/1471-2105-11-395]
-
Schmid, R., et al. (2023) Integrative analysis of multimodal mass spectrometry data in MZmine 3. Nat. Biotechnol. 41: 447-449.
[https://doi.org/10.1038/s41587-023-01690-2]
-
Nothias, L. F., et al. (2020) Feature-based molecular networking in the GNPS analysis environment. Nat. Methods 17: 905-908.
[https://doi.org/10.1038/s41592-020-0933-6]
-
Chen, Q. H., Fu, M. L., Chen, M. M., Liu, J., Liu, X. J., He, G. Q. and Pu, S. C. (2012) Preparative isolation and purification of xanthohumol from hops (Humulus lupulus L.) by high-speed counter-current chromatography. Food Chem. 132: 619-623.
[https://doi.org/10.1016/j.foodchem.2011.10.098]
- Chadwick, L. R. (2004). Estrogens and congeners from spent hops. Ph.D. Dissertation, University of Illinois at Chicago.
-
Stevens, J. F., Taylor, A. W., Nickerson, G. B., Ivancic, M., Henning, J., Haunold, A. and Deinzer, M. L. (2000). Prenylfavonoid variation in Humulus lupulus: distribution and taxonomic significance of xanthogalenol and 4'-O-methylxanthohumol. Phytochemistry 53: 759-775.
[https://doi.org/10.1016/S0031-9422(00)00005-4]
-
Stevens, J. F., Ivancic, M., Hsu, V. L. and Deinzer, M. L. (1997) Prenylflavonoids from Humulus lupulus. Phytochemistry 44: 1575-1585.
[https://doi.org/10.1016/S0031-9422(96)00744-3]
-
Shen, J., Jia, Q., Huang, X., Yao, G., Ma, W., Zhang, H., Ouyang, H. and He, J. (2019) Development of a HPLC-MS/MS method to determine the 13 elements of Semen Cuscutae and application to a pharmacokinetic study in rats. Evid. Based Complement Alternat. Med. 2019: 6518528.
[https://doi.org/10.1155/2019/6518528]
-
Atay, I., Kirmizibekmez, H., Gören, A. C. and Yesilada, E. (2015) Secondary metabolites from Sambucus ebulus. Turk. J. Chem. 39: 34-41.
[https://doi.org/10.3906/kim-1403-47]